青岛能源所与中国医学科学院药物研究所合作揭示不同类型糖基化对蛋白质性质影响的机制
蛋白质的糖基化是最重要的翻译后修饰之一,同时基于蛋白质糖基化的糖工程是对蛋白质和酶进行性质改造的重要工具。不同种类的糖基化修饰对蛋白性质和功能的影响具有显著差异,理解其背后的机制对于蛋白质的糖工程改造具有重要意义。青岛能源所与中国医学科学院北京协和医学院药物研究所合作,通过核磁共振结构解析和分子动力学模拟阐明了为什么糖工程中常用的蛋白质的硫-糖基化不能充分模仿天然的氧-糖基化,揭示了糖苷键类型在形成糖基化功能中的关键作用。该成果在线发表于生物化学领域期刊International Journal of Biological Macromolecules《国际生物大分子杂志》。
蛋白糖基化通过修饰糖基和蛋白间的相互作用,来改善蛋白质的折叠、稳定性、溶解性和生物活性等方面的性能。最常见的天然蛋白糖基化方式是发生在天冬酰胺侧链的氮-糖基化和发生在丝氨酸和苏氨酸侧链的氧-糖基化。而硫-糖基化则是糖基连接在蛋白半胱氨酸侧链的硫原子上,由于硫和氧属于同族元素,许多物理和化学性质相近,并且硫-糖基化在制备过程中具有直接和简便的优点,S-C糖苷键比O-C糖苷键也更加稳定。因此人们认为硫糖基化可以很好的替代氧糖基化,并在一些研究和糖工程开发中得到了应用。然而,另外也有一些研究表明,在糖苷键中从氧到硫的替代会导致糖基化的蛋白具有结构和性质的显著差异,其中的原因并不清楚。
青岛能源所代谢物组学研究组与中国医学科学院药物研究所谭忠平研究员课题组开展了长期的合作,以揭示糖基化对蛋白质结构与性质的影响机制。在前期优化了核磁共振结构计算碳水化合物的参数,实现了解析糖基化修饰蛋白结构的标准流程(PLoS One, 2017, 12:e0189700)的基础上,以里氏木霉的7家族纤维二糖水解酶Cel7A的碳水化合物结合模块(CBM)为模型,从结构水平解释了不同位置的氧-糖基化为什么会对CBM的性质产生完全不同的影响,发现使蛋白性能提高的糖基化作用主要来自于糖基位点附近氨基酸间新的相互作用的引入和蛋白刚性的增加(Biochemistry, 2017, 56:2897-2906)。
为进一步探索不同糖基化对蛋白质结构和性质影响的机制,研究团队以里氏木霉Cel7A的CBM为模型,系统的研究了CBM第三位氨基酸进行不同种类单糖基化和二糖基化修饰的蛋白质(图1)。通过核磁共振结构解析发现,由于S-C糖苷键相比O-C糖苷键具有更长的键长,导致硫-糖基化所连的糖基无法保持氧-糖基化时糖基与蛋白之间的相互作用,在结构中硫-糖基呈现柔性松散的状态,从而大幅削弱了糖基化对于提升蛋白稳定性、增强底物结合的作用。分子动力学模拟的结果同样显示硫-糖基化所连的糖基比氧-糖基化的糖基具有更高的动态性,糖基和蛋白间的氢键强度变化是产生这种动态性差异的关键因素,该动态性改变了结合过程中的焓和熵,影响CBM与底物之间的结合。通过对二糖基化和单糖基化CBM的比较,确定了增加的第二个糖基对蛋白性能影响的结构和动力学机制。该研究揭示了氧-和硫-糖基化之间明显的结构和动力学差异,这些差异能够导致糖基化效应的显著改变,并提示在糖基化改造过程需要谨慎对待糖基化类型的切换,对蛋白质的糖工程研究具有重要的指导意义。
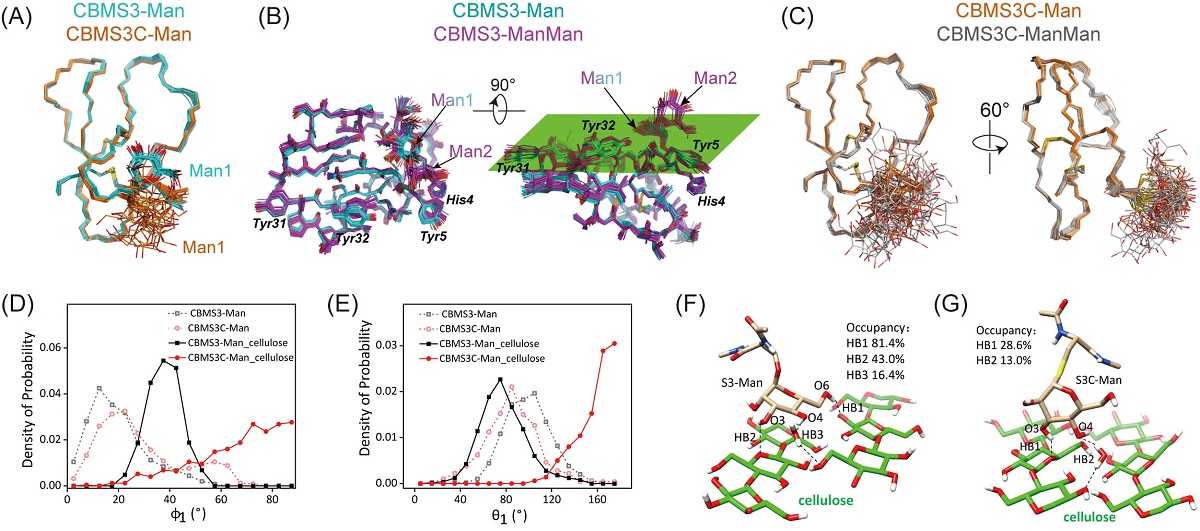
图1 硫-糖基化与氧-糖基化所产生的结构与动力学差异导致了两种糖基化对蛋白质性质的不同影响。(A-C)硫-糖基化的CBM(CBMS3C-Man和CBMS3C-ManMan)与氧-糖基化的CBM(CBMS3-Man和CBMS3-ManMan)的结构差异。硫-糖基化的结构所连接的糖基呈现柔性松散状态而氧-糖基化的结构所连接的糖基呈现为收敛的固定结构。(D-F)分子动力学模拟显示结合纤维素底物后,硫-糖基化的CBM的糖基具有比O-糖基化的糖基更多的运动性和更少的氢键占有率,共同导致其结合底物能力变弱。
青岛能源所代谢物组学研究组陈超副研究员、中国医学科院药物研究所助理研究员马博、青岛能源所蛋白质设计研究组王业飞副研究员为论文共同第一作者,青岛能源所冯银刚研究员、王业飞副研究员和中国医科院药物所谭忠平研究员为论文共同通讯作者。本研究得到了国家重点研发计划、国家自然科学基金委、山东能源研究院、青岛市自主创新重大专项、中国医学科学院医学与健康科技创新工程等项目的资助。(文/图 陈超)。
原文链接:https://doi.org/10.1016/j.ijbiomac.2023.126649
Chao Chen#, Bo Ma#, Yefei Wang#*, Qiu Cui, Lishan Yao, Yaohao Li, Baoquan Chen, Yingang Feng*, Zhongping Tan* (2023) Structural insight into why S-linked glycosylation cannot adequately mimic the role of natural O-glycosylation. Int. J. Biol. Macromol. 126649.
附件下载: